|
|
 发表于 2023-2-27 17:26:02
|
发表于 2023-2-27 17:26:02
|
查看: 311 |
回复: 1
问题1:人类基因组约有30亿个碱基对,DNA甲基化发生在哪儿?熟悉表观遗传的小伙伴答曰:主要是CpG位点。
问题2:人类基因组中有2800万个CpG位点,DNA甲基化发生在哪儿?研究DNA甲基化的小伙伴可能知道,启动子、增强子、genebody上都分布着甲基化。
问题3:这些甲基化如何发生的呢?熟悉DNA甲基化的小伙伴也应该知道,甲基化主要由DNA甲基转移酶(DNMT)负责,具体地分为DNMT1, DNMTA3和DNMT3B。绝大多数DNA甲基化的文献都会告诉你的是,DNMT3A和DNMT3B负责从头甲基化 (de novo methylation),即从无到有。而DNMT1可以识别半甲基化的DNA,即一条链上有甲基化,一条链上没有;在DNA复制的过程中,将对面的那条链也甲基化,即执行DNA甲基化的维持 (methylation maintenance)。
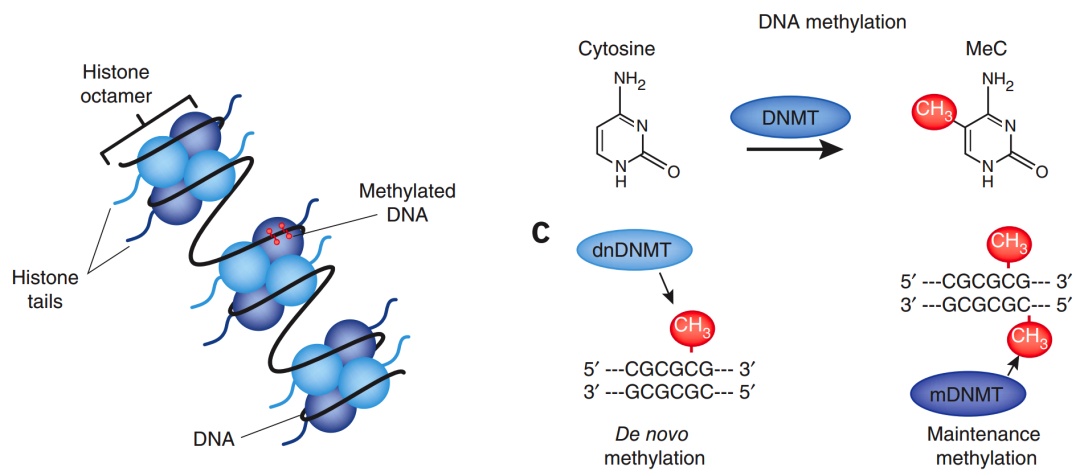
CpG位点的从头甲基化和甲基化维持[1]
接下来的几个问题可能没有那么好回答:为什么我们需要三个DNMT?为什么我们需要两个从头甲基化酶DNMT3A和DNMT3B?基因中的启动子、增强子等调控元件那么多,仅增强子就有100万个,细胞怎么知道在哪些调控元件上添加甲基化,哪些不加?在问题1中没有提到的,甲基化不仅发生在CpG位点,还可以发生在non-CpG位点,比如CH(A/C/T),这些non-CpG的甲基化 (主要富集在神经元中) 是如何发生的呢?在问题2中没有提到的,两个基因之间的区域,即基因间区 (intergenic region) 也有甲基化,基因间区的甲基化是如何形成的呢?这就是今天要讨论的话题。
随着ChIP等技术的普及,人们逐渐观察到:组蛋白修饰可以标记 (mark) 不同的区域,比如H3K27ac/H3K4me1标记活性转录基因的增强子区域,H3K4me3/H3K27ac/H3K9ac标记启动子区域,H3K36me3标记活性转录基因的genebody。
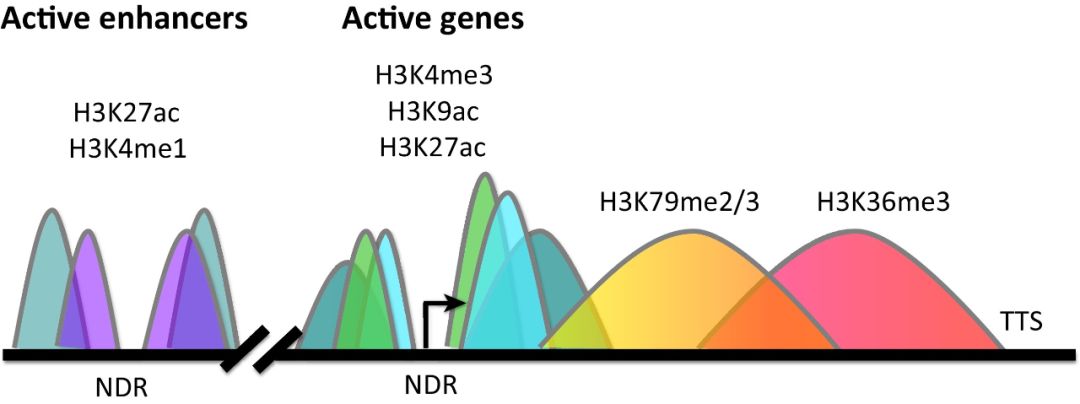
组蛋白修饰标记不同的基因区域[2]
而不同区域的DNA甲基化pattern也与在转录活性密切相关:比如启动子和增强子区域的高甲基化,通常抑制基因的表达;而genebody的高甲基化则与活性转录正相关。组蛋白修饰和DNA甲基化均呈现出特定的pattern,究竟是巧合还是另有其因?如果你看过此前的经典文章《当我们在讨论表观遗传时,究竟在讨论什么?》,应该熟悉这样一个重要论点:组蛋白修饰可以指导DNA甲基化。具体是如何指导的呢?
在2016年的讲座中,颉伟教授就提到:H3K4me3 的存在可以很好地保证基因启动子区域不被 DNA 甲基化。对于 DNMT3A/3B 很重要的 co-factor DNMT3L 无法结合到 H3K4me3 附近的 DNA 上,DNMT3A/3B 就不能过来,即存在一种 active 的机制保证启动子区域不被甲基化[3]。在 genebody 上有 H3K36me3,可以招募 DNMT3B。DNMT3B 里面有一个 domian,可以特异性结合 H3K36me3[4, 5]。最后就形成了一个非常有意思的 pattern,启动子区域没有甲基化,genebody 有甲基化。
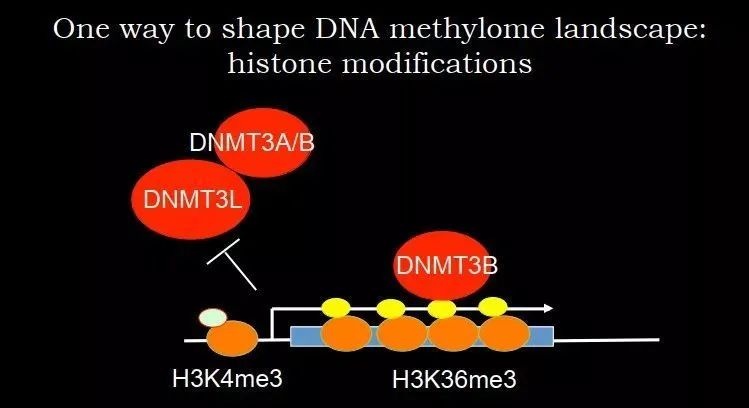
来自颉伟教授2016年讲座七年过去了,这一调控范式不断得到丰富:先是2019年哥伦比亚大学路超、麦吉尔大学Jacek Majewski以及洛克菲勒大学C. David Allis等合作揭示了NSD1介导的基因间区的H3K36me2,可以招募DNMT3A,对基因间区进行甲基化[6]。前几天,圣路易斯华盛顿大学的 Harrison W. Gabel课题组在预印本bioRxiv上发文,揭示NSD1介导的H3K36me2通过招募DNMT3A,对神经元中的non-CpG进行甲基化[7]。

这幅组蛋白修饰指导的DNA甲基化图谱愈发清晰,我梳理如下:
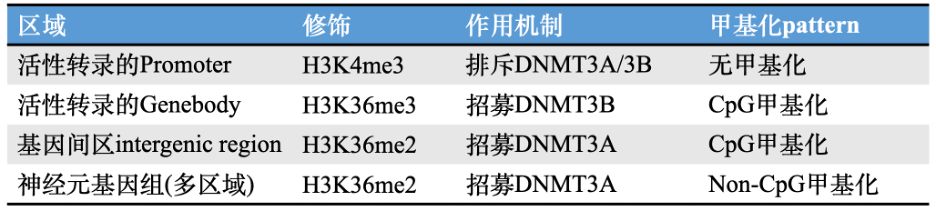
但即便如此,很多问题依旧。组蛋白修饰可以指导DNA甲基化pattern的形成,这一理论中,得先有特定的组蛋白修饰,才能特定有甲基化pattern。
问题1:是否有例外?基因组中一定可以找到只有DNA甲基化,但是没有对应的组蛋白修饰的区域,这些区域的甲基化是如何形成的?当然可以是组蛋白修饰先招募DNMT3A/3B形成,然后再把组蛋白修饰去掉;也可以是压根儿就没有组蛋白修饰什么事情,直接通过其他机制形成了甲基化pattern。
问题2:DNA甲基化能否指导组蛋白修饰pattern的形成?看到相关文献的小伙伴欢迎在留言,感谢!
参考文献:
- Day, J.J. and J.D. Sweatt, DNA methylation and memory formation. Nat Neurosci, 2010. 13(11): p. 1319-23.
- Gates, L.A., C.E. Foulds, and B.W. O'Malley, Histone Marks in the 'Driver's Seat': Functional Roles in Steering the Transcription Cycle. Trends Biochem Sci, 2017. 42(12): p. 977-989.
- Ooi, S.K., et al., DNMT3L connects unmethylated lysine 4 of histone H3 to de novo methylation of DNA. Nature, 2007. 448(7154): p. 714-7.
- Baubec, T., et al., Genomic profiling of DNA methyltransferases reveals a role for DNMT3B in genic methylation. Nature, 2015. 520(7546): p. 243-247.
- Morselli, M., et al., In vivo targeting of de novo DNA methylation by histone modifications in yeast and mouse. Elife, 2015. 4: p. e06205.
- Weinberg, D.N., et al., The histone mark H3K3me2 recruits DNMT3A and shapes the intergenic DNA methylation landscape. Nature, 2019. 573(7773): p. 281-286.
- Hamagami, N., et al., NSD1 deposits histone H3 lysine 36 dimethylation to pattern non-CG DNA methylation in neurons. bioRxiv, 2023: p. 2023.02.17.528965.
|
|